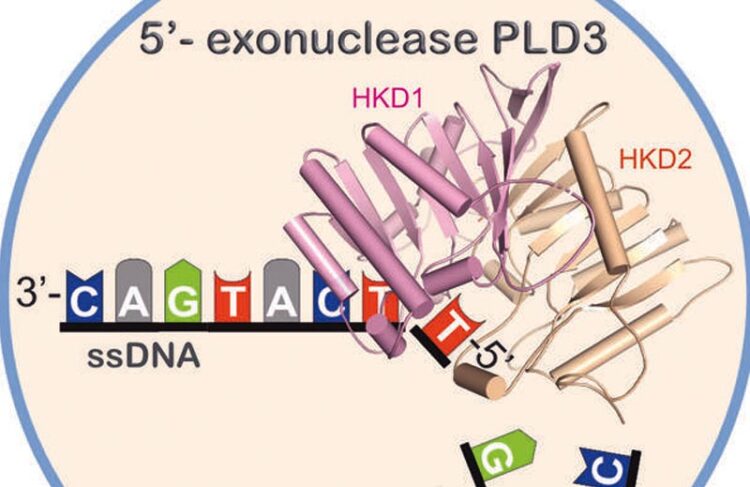
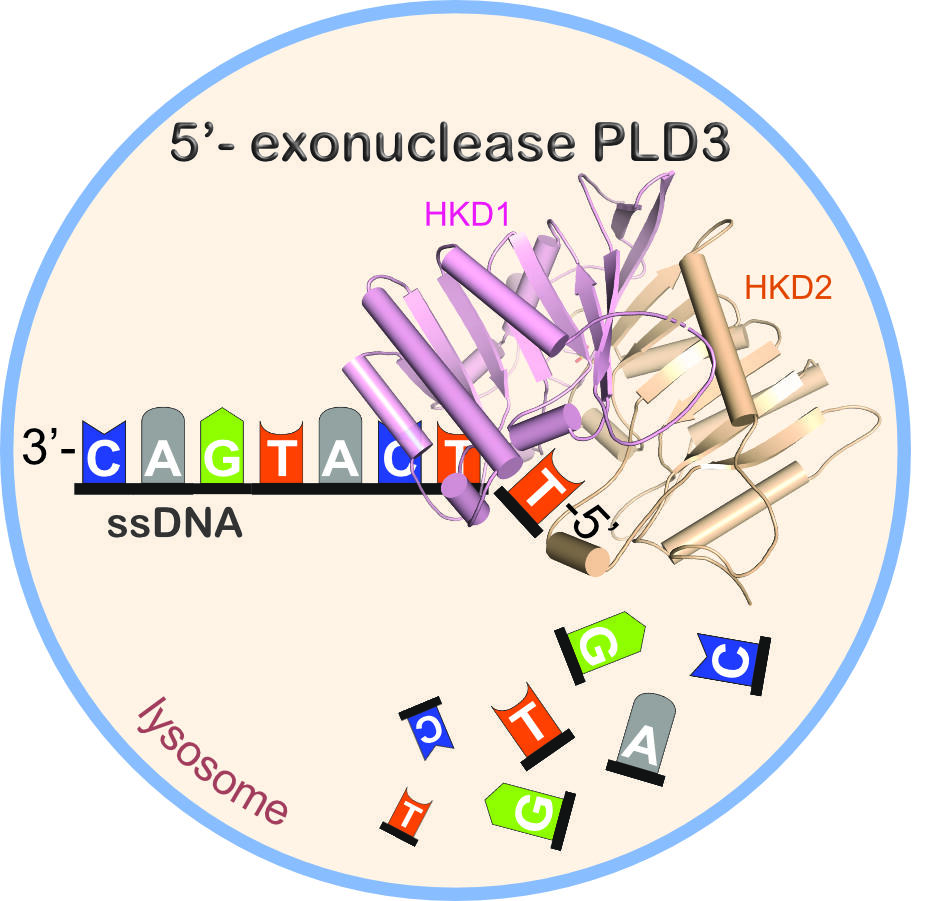
Das Protein PLD3 schneidet von kurzer einzelsträngiger DNA einzelne Nukleinsäure-Bausteine ( A, T, G, C) ab. Dieser Prozess findet innerhalb des katalytischen aktiven Zentrums des Enzyms statt, welches aus zwei strukturellen Untereinheiten (HKD und HKE) besteht.
Der Müll muss aus dem Haus, sonst wird es ungemütlich, gar gesundheitsgefährdend. Ähnliches gilt in unseren Zellen: Nicht benötigte Proteine oder Erbgutstränge sollten beseitigt werden, sonst kann die Zelle und schließlich der gesamte Organismus krank werden. Man vermutet zum Beispiel einen Zusammenhang zwischen Alzheimer und Mutationen, die zu Defekten in der zellulären Müllentsorgung führen. Und Versuche mit Mäusen zeigten: Unterdrückt man den Abbau von DNA und RNA, kann das schwere Autoimmunerkrankungen auslösen.
Doch handfestes Wissen fehlt: „Wie genetische Information in Form von DNA und RNA hergestellt wird, ist beim Menschen gut untersucht. Aber es gibt weniger Erkenntnisse darüber, wie nicht benötigte DNA und RNA wieder abgebaut werden“, erklärt Professor Oliver Daumke, Arbeitsgruppenleiter am Max Delbrück Center. Aus diesem Grund hat er zusammen mit Forschenden der Universität Kiel die Müllabfuhr der Zellen genauer analysiert. Im Mittelpunkt stand dabei das für den Abbau verantwortliche Enzym mit dem Kürzel PLD3. Die Forschenden lösten zunächst seine räumliche Struktur über eine Kristallstrukturanalyse auf und konnten bestimmte Abschnitte identifizieren, die an der Aufspaltung von RNA und DNA maßgeblich beteiligt sind. „So konnten wir den Prozess des Abbaus und die krankmachenden Effekte von Mutationen im PLD3-Protein besser verstehen“, sagt Daumke.
Mutationen im PLD3-Gen steigern Alzheimerrisiko
PLD3 gehört zu einer Proteinfamilie von Enzymen, die in menschlichen Zellorganellen – den Lysosomen – zelluläre Fette zerlegt. Für die Herstellung von PLD3 im menschlichen Körper ist das zugehörige Gen mit demselben Namen verantwortlich. „Wir haben uns schon länger mit dem PLD3-Gen beschäftigt, weil es seit einigen Jahren Hinweise darauf gibt, dass Mutationen in diesem Gen bei der Entstehung von Alzheimer eine Rolle spielen“, sagt Professor Markus Damme, an der Christian-Albrechts-Universität zu Kiel. „Unsere Arbeiten und die von anderen Forschenden offenbarten dann in den vergangenen Jahren, dass PLD3 offenbar anstelle von Fetten DNA und RNA abbaut“, sagt Damme.
„Wie dies gelingt, war aber unklar“, erklärt Cedric Cappel, Wissenschaftler in Dammes Gruppe und Erstautor der Publikation. „Die Idee war daher, sich die Struktur des Proteins genauer anzusehen – um so vielleicht etwas über die Verbindung zu Alzheimer herauszufinden.“ Cappel stellte also das Protein her und schickte es der Strukturbiologin aus Daumkes Arbeitsgruppe Dr. Yvette Roske, ebenfalls Erstautorin der Veröffentlichung. Ihr gelang es, winzige Kristalle von PLD3 herzustellen. Bestrahlt man diese mit Röntgenstrahlen, entsteht ein Beugungsmuster, mit dem sich die Struktur des Proteins rekonstruieren lässt. So konnte Roske schließlich die Kristallstruktur mit und ohne eine gebundene RNA darstellen und analysieren. „Wir haben herausgefunden, dass sich zwei dieser Proteine zu einem sogenannten Dimer zusammenlagern. Das kennen wir von anderen Enzymen aus dieser Familie nicht“, berichtet sie. Aber weshalb ist das nötig? „Es könnte sein, dass das Protein nur im Doppelpack stabil ist“, vermutet Cappel, „ansonsten wird es wahrscheinlich abgebaut.“
Die Arbeit der beiden Forschungsgruppen liefert den ersten strukturellen Beweis für den DNA- und RNA-Abbau durch PLD3. „Wir können nun erstmalig den ungefähren Reaktionsmechanismus nachvollziehen“, sagt Roske. Darüber hinaus fanden die Forschenden zwei Stellen im Protein, die für dessen Funktion wichtig und mutmaßlich in Alzheimerpatienten verändert sein können – ein erster Hinweis auf einen möglichen krankmachenden Mechanismus.
„Unsere Arbeit liefert eine Landkarte für das Protein“, erklärt Cappel. Zukünftige Studien über PLD3 können diese nutzen, um etwa folgende Fragen zu klären: „Welche Stellen sind für die Funktion von PLD3 notwendig, und was passiert, wenn ich daran etwas ändere?“ Die Forschenden hoffen, dass man so genauer versteht, welche Rolle das Protein bei bestimmten Krankheiten spielt. Und womöglich lässt sich auf Basis dieses Wissens dann korrigierend eingreifen.
Literatur
Yvette Roske, et al. (2023): “Structural analysis of PLD3 reveals insights into the mechanism of lysosomal 5’-exonuclease-mediated nucleic acid degradation”; in Nucleic Acids Research, DOI: 10.1093/nar/gkad1114
Kontakte
Christina Anders
Redakteurin, Abteilung Kommunikation
Max Delbrück Center
+49 (0)30 9406 2118
christina.anders@mdc-berlin.de oder presse@mdc-berlin.de
Max Delbrück Center
Das Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft (Max Delbrück Center) gehört zu den international führenden biomedizinischen Forschungszentren. Nobelpreisträger Max Delbrück, geboren in Berlin, war ein Begründer der Molekularbiologie. An den Standorten in Berlin-Buch und Mitte analysieren Forscher*innen aus rund 70 Ländern das System Mensch – die Grundlagen des Lebens von seinen kleinsten Bausteinen bis zu organ-übergreifenden Mechanismen. Wenn man versteht, was das dynamische Gleichgewicht in der Zelle, einem Organ oder im ganzen Körper steuert oder stört, kann man Krankheiten vorbeugen, sie früh diagnostizieren und mit passgenauen Therapien stoppen. Die Erkenntnisse der Grundlagenforschung sollen rasch Patient*innen zugutekommen. Das Max Delbrück Center fördert daher Ausgründungen und kooperiert in Netzwerken. Besonders eng sind die Partnerschaften mit der Charité – Universitätsmedizin Berlin im gemeinsamen Experimental and Clinical Research Center (ECRC) und dem Berlin Institute of Health (BIH) in der Charité sowie dem Deutschen Zentrum für Herz-Kreislauf-Forschung (DZHK). Am Max Delbrück Center arbeiten 1800 Menschen. Finanziert wird das 1992 gegründete Max Delbrück Center zu 90 Prozent vom Bund und zu 10 Prozent vom Land Berlin.
Originalpublikation:
Yvette Roske, et al. (2023): “Structural analysis of PLD3 reveals insights into the mechanism of lysosomal 5’-exonuclease-mediated nucleic acid degradation”; in Nucleic Acids Research, DOI: 10.1093/nar/gkad1114
Weitere Informationen:
https://www.mdc-berlin.de/de/daumke
https://www.uni-kiel.de/Biochemie/scripte/dynamic/groups/damme/group.php